表观遗传学整体实验1、提供表观遗传学整体实验方案:客户根据课题标书及需做实验的主体内容,拟定并提供初步的项目方案,含课题申请书,实验方案,实验设计,实验思路,实验文献等与实验相关等内容和资料。或者由嘉美实验协助实验委托者完成表观遗传学整体实验方案的设计。
2、项目评估报价:公司技术团队根据客户提供的相关资料,综合评估项目可行性,与客户共同确定准确的实验项目执行方案,给出准确的实验价格。嘉美实验协助设计的课题直接给出准确的报价。
3、签订服务协议:签署服务协议并预付部分费用,做好实验初步准备及项目预安排。
4、执行服务项目:正式启动实验进程,全面展开实验,确保项目有效执行。
5、沟通项目进展:及时沟通反馈实验的Newest进展,随时了解掌控实验的Newest动态。
6、完成项目交付:支付剩余尾款,交付实验结果,正确应用实验结果,提供售后服务。

一、癌症样本的表观遗传学分析
癌症表观遗传学的研究,如甲基化异常和转录因子结合的改变,能够帮助人们深入了解重要的致癌通路。由于甲基化改变往往激活或沉默基因,表观基因组的变化可能影响基因表达和癌症发展的速度。
新一代测序(NGS)和芯片技术可检测癌症中甲基化模式的改变及其他表观遗传学改变。
二、通过测序了解癌症表观遗传学
甲基化异常是癌症中一种常见的表观遗传学改变。甲基化的NGS分析让研究人员能够通过甲基化基因组的测序而鉴定和追踪甲基化模式。
1、甲基化测序简介
胞嘧啶甲基化可能明显改变基因的时空表达和染色质重塑。全基因组重亚硫酸盐测序(WGBS)利用新一代测序(NGS)和全基因组分析的力量,提供整个基因组甲基化模式的单碱基分辨率全面视图。
2、甲基化测序的优势
1)可发现整个基因组中所有CpG、CHH和CHG区域的甲基化模式
2)能以少量的DNA捕获全面的样品多样性
3)能查看大多数物种基因组中几乎每个胞嘧啶的甲基化

3、测序整个样品 – 而不丢失信息
许多方法利用NGS的高质量和灵敏度来进行甲基化分析。大多数方法依赖于DNA的重亚硫酸盐转化来检测未甲基化的胞嘧啶。在文库制备过程中,重亚硫酸盐将未甲基化的胞嘧啶转化成尿嘧啶。转化后的碱基在测序中被识别为胸腺嘧啶(PCR后),并通过序列计数来确定甲基化胞嘧啶的比例。
重亚硫酸盐转化测序可通过靶向方法完成,如扩增子甲基化测序(Methyl-seq),或全基因组重亚硫酸盐测序(WGBS)。此外,TAB-Seq和OxBS等替代方法也利用NGS来鉴定羟甲基化(5-hmC)并开展甲基化(5-mc)分析。
4、甲基化测序的流程
1)文库制备:构建全基因组重亚硫酸盐测序文库,仅需50 ng DNA。
2)测序
3)数据分析和储存
三、癌症表观遗传学的芯片分析
甲基化芯片可平行分析多个样品,实现了全表观基因组关联研究。芯片定量检测基因组内的甲基化位点,帮助人们了解癌基因和通路的调控。
1、甲基化芯片简介
DNA甲基化对基因表达调控起着动态的重要作用。 借助MethylationEPIC BeadChip,研究人员可以基于单核苷酸分辨率定量检测基因组中的850,000多个甲基化位点。包括FFPE在内的多重样本可以并行分析,以便在每样本成本的情况下实现高通量功能。
MethylationEPIC BeadChip是全表观基因组关联研究(EWAS)的理想之选。它提供了由专家精选的全面的覆盖范围,囊括99%的RefSeq基因,95%的CpG岛,高覆盖度的增强子区域和其他内容类别。超过90%的HumanMethylation450K位点内容都涵盖在内。
2、甲基化芯片的优势
MethylationEPIC BEadChip兼具全面的覆盖范围和高通量功能,因此是EWAS的理想之选。其他优势包括:
1)全面的全基因组覆盖范围(> 850,000个甲基化位点)
CpG 岛
非CpG和差异甲基化位点
FANTOM5增强子
ENCODE染色质
ENCODE转录因子结合位点
miRNA启动子区域
2)实验分析方法重现性
98%(针对技术性重复)
98%(针对传统HumanMethylation450K芯片对照,MethylationEPIC芯片采用的相同样本)
>90%(针对HumanMethylation450K位点内容)
3)用户友好的简化工作流程
4)与FFPE样本兼容

3、甲基化芯片工作流程
MethylationEPIC BeadChip遵循用户友好的简化的工作流程,可以从低样本起始量(低至250 ng)同时处理多达96个样本。
该甲基化芯片针对常规样本和FFPE样本提供单CpG位点级别的定量测量,因此能实现超级精细的解析度,方便用户了解表观遗传的变化。
1)样品的制备:MethylationEPIC BeadChip试剂盒运用一代和二代Infinium实验分析方法化学技术,支持无PCR和低样本输入量(低至250 ng)。
2)阵相处理和扫描:iScan 系统可迅速准确地扫描成百上千个样本,支持高通量的BeadChip处理。
3)数据分析:GenomeStudio软件和甲基化模块易如反掌的差异甲基化分析,并显示染色体坐标、GC百分比、CpG岛的位置和甲基化ß值。
四、检测肿瘤中的DNA-蛋白质相互作用
转录因子结合的改变是癌症中一种普遍的表观遗传模式。染色质免疫沉淀测序(ChIP-Seq)实验可提供与感兴趣的蛋白质相关联的DNA区域的完整写照。
1、ChIP-Seq简介
通过染色质免疫沉淀(ChIP)检测和测序技术相结合,ChIP测序(ChIP-Seq)成为鉴定转录因子及其他蛋白因子在全基因组范围内的DNA结合位点的强有力方法。在ChIP操作之后,通过特异抗体将DNA结合蛋白免疫沉淀。结合的DNA被同时沉淀、纯化并测序。
新一代测序(NGS)在ChIP上的应用已经揭示了一些在各种疾病和生物学通路(如发育和癌症发展)中发挥作用的基因调控事件的线索。ChIP-Seq能够在全基因组规模上实现对蛋白与核酸相互作用的全面检测。
2、ChIP-Seq的优点
1)可在任何生物体的整个基因组中捕获转录因子或组蛋白修饰的DNA靶点
2)确定转录因子的结合位点
3)结合RNA测序和甲基化分析,揭示基因调控网络
4)可兼容不同起始量的DNA样品

3、无偏向性地研究表观遗传模式
ChIP-Seq可鉴定DNA结合蛋白的结合位点,可用于定位一个给定蛋白的整体结合位点。芯片及其他研究表观基因组的方法往往有着内在的偏向性,因为它们需要来自已知序列的探针,ChIP-Seq则不同,不需要任何先验知识。ChIP-Seq通过大规模并行测序提供全基因组的图谱,产生覆盖多个样品的数百万次计数分析,实现经济且精确的分析。


4、ChIP-Seq的流程
应用Illumina的边合成边测序(SBS),该技术是GlobalZ广泛采用的NGS技术,已产生了Global大约90%的测序数据。
1)文库制备
2、测序
4)数据分析
 表观遗传学整体实验
表观遗传学整体实验
 061.【表观遗传-m6A深度研究】绝招在身,走遍天下!掌握表观遗传学前沿产品研究工具!
061.【表观遗传-m6A深度研究】绝招在身,走遍天下!掌握表观遗传学前沿产品研究工具!
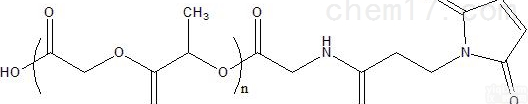 1 帕比司他 CAS号:404950-80-7 表观遗传学
1 帕比司他 CAS号:404950-80-7 表观遗传学
 ZYMO甲基化表观遗传学系列
ZYMO甲基化表观遗传学系列
 表观遗传学 Goat F(ab’)2 Anti-Rabbit IgG(H+L), ...
表观遗传学 Goat F(ab’)2 Anti-Rabbit IgG(H+L), ...
 表观遗传学 Goat Anti-Rabbit IgG(H+L), Human ad...
表观遗传学 Goat Anti-Rabbit IgG(H+L), Human ad...
 表观遗传学 Goat Anti-Rabbit IgG(H+L), Mouse/Hu...
表观遗传学 Goat Anti-Rabbit IgG(H+L), Mouse/Hu...
 表观遗传学 Goat Anti-Rabbit IgM-PE抗体
表观遗传学 Goat Anti-Rabbit IgM-PE抗体
 表观遗传学 Goat Anti-Rabbit IgG Fab-UNLB抗体
表观遗传学 Goat Anti-Rabbit IgG Fab-UNLB抗体
 表观遗传学 Goat Anti-Rabbit IgG-PE抗体
表观遗传学 Goat Anti-Rabbit IgG-PE抗体
 表观遗传学 Goat Anti-Rabbit IgG-BIOT抗体
表观遗传学 Goat Anti-Rabbit IgG-BIOT抗体
 表观遗传学 Goat Anti-Rabbit IgG-AP抗体
表观遗传学 Goat Anti-Rabbit IgG-AP抗体